京・富岳やゲノム研究を開発推進した元国立研究開発法人理化学研究所(以下、理研という)の研究チームで構成される「先端加速システムズ(株)・(株)ダナフォーム・順天堂大学の共同研究グループ」は、ヒト全ゲノム解析を、10分以下(最短記録7分39秒)で終了する汎用パソコンやCPUサーバー用のソフトウエアシステム(AAS-G1)を開発した。ゲノムシーケンサーから排出されるヒト全ゲノムの30倍の配列をヒト全ゲノム標準配列にアラインメントし、変異の位置を特定する一次解析は、従来は最高速のものでも、高価な専用計算機や大型スパコン(FPGAやGPU搭載)等の特殊なハードウエアを使って15分~30分で行われていた。これらの従来システムは、通常、共用で使われてきたが、今回のソフト開発により、個々の解析者が個人使用の安価な汎用パソコン(CPU搭載)を用いて、日常的に10分を切る超高速解析が実現されることになる。
ヒトゲノムシーケンスデータは、その個体の遺伝的背景から疾病発症の予測、ガンの薬剤応答性などの医療応用に幅広く使われている。従来は、ゲノム情報を引き出す手法として、遺伝子の変異箇所のみの塩基を決定するタイピングや、特定の遺伝子領域のみを増幅したり、濃縮したりして塩基配列を決定するパネルシーケンスなどが使われてきた。近年、ヒト全ゲノム解析は、全ゲノムシーケンスのデータ産出コストが年々安価になっているうえ、ゲノムの各部分のタイピングやパネルシーケンスよりもはるかに多くの情報量を含むため、全ゲノムシーケンスがタイピングやパネルシーケンスに置き換わり、主流を占めるであろうという予想がなされていた。
しかし、シーケンスコストが下がる一方、出てくる大量の断片化されたシーケンスをつなぎ合わせ、変異場所を見つけるという一次解析に、膨大な計算機資源を要することが、全ゲノムシーケンスが、従来法に一気に置き換わらない一つの要因であった。従来、この膨大な計算を行うため、特殊なハードウエア(FPGA、GPUなど)が用いられていたが、これらの設備が高価であること、共用施設として利用される等、一般普及におのずと限界があった。
京・富岳やゲノム研究を開発推進した元理研の研究チームで構成される「先端加速システムズ(株)・(株)ダナフォーム・順天堂大学の共同研究グループ」が完成したソフトウエアシステム(AAS-G1)により、高速計算機で従来15分から30分かかっていたヒト全ゲノムの一次解析を、個々のユーザーが個人使用のパーソナルコンピューターやCPUサーバーで10分以内に(最短時間が7分39秒)実行される事が可能となった。これにより、近い未来にはシーケンシング法の高速化とともに、全ゲノムの情報解析の高速化と低コスト化のための安価で大量の計算機資源の供給が可能となった。ヒトゲノムの迅速な診断が必要な、「新生児の全ゲノム診断」や、「1泊人間ドック」などの医療応用におけるヒト全ゲノム解析の高速化のみならず、全世界のゲノムプロジェクトにおけるシーケンスデータの解析を、安価に高速に行える計算機資源が提供されるようになり、ゲノムプロジェクト全体の底上げに大きく貢献することが期待される。
1.ゲノム解析の超高速パソコンソフトウエアAAS-G1の特徴
(1)超高速解析
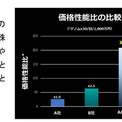
AAS-G1は超高速でヒトゲノムのアラインメントと変異検出が可能な遺伝子解析ソフトウエアである。シーケンサーから排出される断片化ゲノム配列をつなぎ合わせる(アライメント)作業と変異箇所を抽出する(バリアントコール)作業の双方(1次解析)を短時間に解析(F‐measure:0.989)できる。多重度(Deepness)30×の断片化ゲノム配列(全部で900億塩基)をつなぎ合わせ、標準配列と比べて変異箇所を抽出する作業が、10分以下(最速7分39秒)で実行できる。従来の他社解析ツールは、特殊なハードウエア(FPGAやGPU)を用いたものであり、同じ断片化シーケンスデータの解析速度は、15分から30分であった。

(実施例)
最新のインテル互換CPU(AMD 64-core EPYC 9554x2)のみ(FPGAやGPUなし)を用いてヒト全ゲノム解析(x30、アライメントとバリアントコール)を10分以下(最短時間7分39秒)で実行した。業界標準のBWA-MEM+GATKでは20時間以上かかっていた。
(2)変異検出の正確性が高くすぐれたパーフォーマンス
米国FDAが実施しているGolden Standardのゲノム配列を利用したゲノム1次解析の正確性評価システム(Precision FDA truth challegeV2 https://precision.fda.gov/challenges/10) のデータを用いた正確性スコア(F1 Score)は、99.0%であり、(F‐measure:0.989)、精度はBWA-MEM+GATKを用いたものときよりも高水準だった。

(3)パーソナルコンピューター(CPU)で稼働するシステム
このソフトを使用するハードウエアは、CPUを搭載したパーソナルコンピューターであればどの機種でも稼働する。PCを稼働用ハードとするため、非常に安価である。他の特殊なハードウエア(FPGAやGPUなど)に依存した従来のゲノム解析システムを一層高速化しようとすれば、これらのハードウエアの更なる開発に膨大なコストがかかるのに比し、AAS-G1は、ソフトウエア自体の改良で高速化が実現可能であるのみならず、本システムが世界で汎用的に使われているCPUを搭載するパーソナルコンピューターやサーバーで稼働するため、モーア法則に従いCPUが高速化するに比例して、本ソフトウエアの解析速度が増すという特徴を有す。
2.AAS-G1のゲノム解析システム進化上の意義
パソコン(CPU)で稼働する安価な超高速ゲノム解析ツールが、利用可能になったために、あたかも、固定電話から携帯電話ができたときのように、ゲノム解析が、スパコン用などの特殊なハードウエア(FPGAやGPUなど)から汎用パソコン稼働となり、さらに高速化しているため、解析者一人1台の計算機資源を割り当てるほうが経済効率もはるかに高くなった。また、一方、ゲノムセンターでは、安価な超高速ゲノム解析ツールが利用可能になったことで、従来の高速計算機による解析システムと比べ、同じコストで多数のPCを並列に稼働させることができるようになった。ヒトゲノム解析の計算機資源が革新的に拡大できる基礎ができたといえよう。ゲノム解析は、ゲノムシーケンスコストが指数関数的に安価になったこととあいまって、ゲノム情報の抽出において、タイピングやパネルシーケンスから全ゲノムシーケンスへの移行がますます加速化される。下記に述べるヒト全ゲノムの医療応用のルーチン化が一気に近づいたと言える。
3.AAS-G1の原理
AAS-G1では、このアライメント部分に新しく開発したアルゴリズム(特許出願中)を使い、バリアントコール部分についても最新のCPUに対応した最適化を行なうことで全体として 100倍以上の高速化を達成しました。
4.AAS-G1の医療応用
ゲノムシーケンスコストが下がっている現在の環境下で、全ゲノム解析に必要な計算機資源のコストが下がったことにより、ヒト全ゲノム情報に依存する医療が、身近になったと言える。AAS-G1は、「順天堂大学大学院医学研究科 難病の診断と治療研究センター」に導入され、30Xヒトゲノムシーケンスのアセンブルとバリアントコールが8分25秒で完了した(最短時間7分39秒)。このシステムを用いて将来的には、順天堂大学医学部附属 順天堂医院 周産期センターでは「新生児のゲノム解析による疾患同定」や、同医院 総合診療科における「一日人間ドックの全ゲノム解析による疾患予測メニュー」等の道が開けることになった。
5.本ソフトウエアAAS-G1の販売開始について
(1)製品名:「AAS-G1」
(2)本ソフトウエアを搭載したハードウエアシステムは、下記日本バイオインフォマティクス学会年会の発表時より、ナベインターナショナル、プラナスソリューションズなどのハードウエアベンダーから発売される。また、ソフトウエア販売も、(株)ダナフォームより開始される。
(3)本製品は、2023年9月8日10時50分~12時20分 日本バイオインフォマティクス学会年会(第12回生命医薬情報学連合大会IIBMP2023)で発表される。
演者:先端加速システムズ(株) 代表取締役 姫野龍太郎
場所:柏の葉カンファレンスセンター(〒277-0871 千葉県柏市若柴178-4 三井ガーデンホテル 柏の葉 ホテル&レジデンス棟2階)
●AAS-G1の開発グループについて
京・富岳やゲノム研究を開発推進した元理研の研究チームで構成される先端加速システムズ(株)(姫野龍太郎代表取締役)・(株)ダナフォーム(林崎良英代表取締役)・順天堂大学(新井一学長)の共同研究グループ研究チームのメンバー(牧野淳一郎)により、AAS-G1は開発された。本システムを、順天堂大学大学院医学研究科 難病の診断と治療研究センター(岡崎康司センター長)が導入し、その臨床応用を推進している。
1.牧野淳一郎
計算機科学の権威。1985年東京大学卒業後、1990年博士号取得。その後東京大学助教授を経て、国立天文台教授、東京工業大学教授を経て、2012年から2022年理研計算科学研究機構エクサスケールコンピューティング開発プロジェクト 副プロジェクトリーダーを経て、現神戸大学教授、先端加速システムズ(株)を創設し、取締役に兼務就任。高速計算機開発分野のノーベル賞と呼ばれているGordon Bell賞を7回受賞。特に、高速計算機の演算素子NM Core1とNM Core2を開発し、単位エネルギー当たりの演算速度を競うGreen500で2021年と2022年世界1位(Gold Medal)を獲得。Natureをはじめとするトップジャーナルに188報発表。
2.姫野龍太郎
大型計算機システムの第一人者。1977年京都大学卒業後、1979年日産自動車中央研究所入社、自動車の流体力学を計算機シミュレーション研究でシニアリサーチャー、1998年東京大学教授、埼玉大学助教授、2004年理研情報基盤センター長として大型計算機センターの企画設立運営を行う。2006年理研次世代スーパーコンピュータ開発実施本部グループディレクターとして、京・富岳やゲノム研究を開発推進、2020年先端加速システムズ(株)代表取締役に就任、2022年より順天堂大学健康データサイエンス学部特任教授、現在に至る。2005年文部科学大臣賞、2006年高速計算機開発分野のノーベル賞と呼ばれているGordon Bell賞を受賞。自動車、ボール(野球)などの流体力学シミュレーション領域等で、74報発表
3.林崎良英
トランスクリプトミクス(RNA)などのオミックス科学の第一人者、1982年大阪大学医学部卒、医師、医学博士、1998年理研ゲノム科学総合研究センタープロジェクトディレクター、2008年理研オミックス基盤研究領域長、2013年理研予防医療プログラム長、2012年より順天堂大学客員教授兼務を経て、2021年(株)ダナフォーム代表取締役、現在に至る。国際FANTOMプロジェクトを創始し、国際標準オミックスデータベース作成で世界をリード。これを用いて、ノーベル賞受賞者山中伸弥博士のiPS細胞が開発される。スウェーデン王立カロリンスカ研究所客員教授、クイーンズランド大学名誉教授。2004年文部科学大臣賞、2007年紫綬褒章、2012年カロリンスカ研究所、名誉博士号、2013年国際ヒトゲノムコンソーシアム、Chen賞、2019年欧州生物学機構(EMBO)Associate Member等。Nature Science等の国際誌に575報発表
4.岡崎康司
オミックス医学、ゲノム医学の第一人者。ミトコンドリア疾患等、多数のヒト疾患に焦点をあて、オミックス医学を推進している。1986年岡山大学医学部卒、循環器病専門医、臨床遺伝専門医。1995年大阪大学医学部大学院博士課程修了、医学博士、1999年理研ゲノム科学総合研究センター、チームリーダー、2003年埼玉医科大学ゲノム医学研究センターゲノム科学部門部門長教授、2008年同センター所長、2017年順天堂大学大学院医学研究科 難治性疾患診断・治療学教授、難病の診断と治療研究センター センター長、教授。2018年から理研 生命医科学研究センター 応用ゲノム解析技術研究チーム チームリーダー兼任。2001年 人間力大賞グランプリ、経済産業大臣奨励賞。Natureを含む国際誌に321報発表。
●用語説明
1.アラインメント
シーケンスによって得られた短い断片的な配列がゲノムのどの部分に当たるかを決定する計算工程。リファレンスとなるヒトゲノム標準配列に断片配列を当てはめて行う。数億本の断片配列について、ゲノム配列全てを検索する必要があるため、膨大な計算量が必要となる。

2.パソコン
ここでは、特定の用途に特化したFPGAやGPUを搭載せず、汎用機として運用されるコンピューターを指す。
3.CPU (Central Processing Unit)
日本語では「中央演算処理装置」と訳される、コンピューターの全体を制御するための半導体チップ。数値計算を行う際にも用いられるため、CPUの性能は計算速度に大きな影響を与える。
4.タイピング
ゲノム中の特定の場所が個体により異なる部分(変異箇所、バリアント)がある場合、ゲノム中の特定の場所のみに注目し、各個体のゲノムの、その特定の位置に、どのパターンの変異があるのかだけを決定していく解析方法。
5.パネルシーケンス
ゲノム内の標的領域のみをシーケンスするアプローチのこと。
標的領域を絞る方法として、標的領域のみをPCR増幅するアンプリコン法と、チップの上に標的領域のみをハイブリダイズさせ、補足することで濃縮するキャプチャー法がある。
従来は、シーケンスコストが高かったために、シーケンスする領域のみを絞ったパネルシーケンスしたほうが、全ゲノムシーケンスよりシーケンスコストが安くつき、解析するべき情報が少ないため情報解析の計算機資源も小規模安価で済むという評価が下されていた。しかし、近年、標的領域を濃縮するためのアンプリコン法の増幅試薬に係る値段や、ハイブリダイゼーションのためのチップの値段のほうが、シーケンスコストより高くつき、さらに、全ゲノムシーケンスのほうがライブラリーの作成がはるかに簡単で迅速であり、一回の実験で得られる情報量もはるかに多いので、パネルシーケンスよりも全ゲノムシーケンスに解析戦略が転換しつつある。そのうえ、今回のAAS-G1の出現により、解析の計算機資源もはるかに安価になったために、あらゆる意味で全ゲノム解析がパネルシーケンスよりも科学的に安価で有意義なデータが出せるため、パネルシーケンスに置き換わりつつある。

6.FPGA (Field Programmable Gate Array)
ある特定の計算を効率的にこなせるような命令をユーザーが物理的に組み込むことができる半導体チップ。CPUしか用いない一般的なソフトウエアよりも速い計算が可能となる。CPUやGPUはどんな処理でもこなせるよう、汎用的な「命令セット」を備えるのに対し、FPGAはプログラムが物理的な回路となって実行される。
7.GPU (Graphics Processing Unit)
3次元グラフィクスの処理に特化した半導体チップで、CPUの性能を補う形で用いられる。近年ではその数値計算能力の高さから、ゲノム解析や機械学習を含めた各種の科学計算に広く用いられている。
8.バリアントコール
検体から変異箇所を抽出する計算工程。得られた検体DNAの塩基配列をリファレンスゲノム配列と比較し、リファレンスゲノムと配列が異なる部分を変異として検出する。

9.F‐measure
バリアントコールの正確性と網羅性を評価するための指標。1.0に近づくほど正確性と網羅性のバランスが取れていると判定される。正確性に関する指標である適合率(全陽性判定中の擬陽性の割合)と、網羅性に関する指標である再現率(真の陽性のうち、実際に陽性判定されたものの割合)の調和平均として算出される。
10.多重度(Deepness)
ゲノム上の各塩基が平均何回されるかの指標。シーケンスの塩基配列読み取り精度は100パーセントではなく、エラーによる読み間違いが生じる。同じ配列を何度も読み取り、比較することで、読み間違いを認識することができる。この平均読み取り回数を多重度と呼ぶ。多重度30×は、ゲノムの各塩基が平均30回シーケンスされることを意味し、ゲノム解析のスタンダードになっている。

11.AMD (Advanced Micro Devices, Inc.)
アメリカの半導体メーカー。Intel社やNvidia社と並ぶような性能を持つ CPU/GPU をラインナップに持つ。最近では FPGA にも力を入れている。
12.BWA-MEM
シーケンサーから出力された配列断片をゲノムの標準配列(リファレンスゲノム)に照合、アライメントするソフトウエア。2009年に発表され、ゲノム解析でのスタンダードソフトウエアとして用いられている。バローズ・ホィーラー変換という手法を用いる。メモリーの使用量を抑えており、メモリー容量が少ないPCでも実行できるが、実行時間が24時間近くもかかるのが問題になっている。
13.GATK
変異検出を行うためのソフトウエア。米国Broad研究所によって開発された。2010年に発表され、ゲノム解析でのスタンダードソフトウエアとして用いられている。
14.ショットガンシーケンス
ゲノム配列を決定するために使用される手法。ゲノムDNAをランダムに物理的に切断し、得られたDNA断片をシーケンスして配列を決定、DNA断片の重なり合った部分をコンピューターによってつなぎ合わせることで、連続した全ゲノムの遺伝子配列を決定する(図1参照)。
配信元企業:学校法人 順天堂



コメント